【设计|从头设计路线,我国科学家建立蛋白质设计新方法】IT之家 2 月 10 日消息 , 据中国科大官网 , 中国科学技术大学刘海燕教授、陈泉副教授团队采用数据驱动策略 , 开辟出一条全新的蛋白质从头设计路线 。
IT之家了解到 , 相关成果以“用于蛋白质设计的以主链为中心的神经网络能量函数”为题于北京时间 2 月 10 日发表于 Nature 。
据介绍 , 蛋白质是生命的基础 , 是生命功能的主要执行者 , 其结构与功能由氨基酸序列所决定 。 目前 , 能够形成稳定三维结构的蛋白质 , 几乎全部是天然蛋白质 , 其氨基酸序列是长期自然进化形成 。 在天然蛋白结构功能不能满足工业或医疗应用需求时 , 想要得到特定的功能蛋白 , 就需要对其结构和序列进行设计 。
中国科学技术大学相关团队长期深耕计算结构生物学方向的基础研究和应用基础研究 。 刘海燕教授、陈泉副教授团队十余年来致力于发展数据驱动的蛋白质设计方法 , 建立并实验验证了给定主链结构设计氨基酸序列的 ABACUS 模型 , 进而发展了能在氨基酸序列待定时从头设计全新主链结构的 SCUBA 模型(图 1) 。 SCUBA 采用了一种新的统计学习策略 , 基于核密度估计(或近邻计数 , NC)和神经网络拟合(NN)方法 , 从原始结构数据中得到神经网络形式的解析能量函数 , 能够高保真地反应实际蛋白质结构中不同结构变量间的高维相关关系 , 在不确定序列的前提下 , 连续、广泛地搜索主链结构空间 , 自动产生“高可设计性”主链 。
文章图片
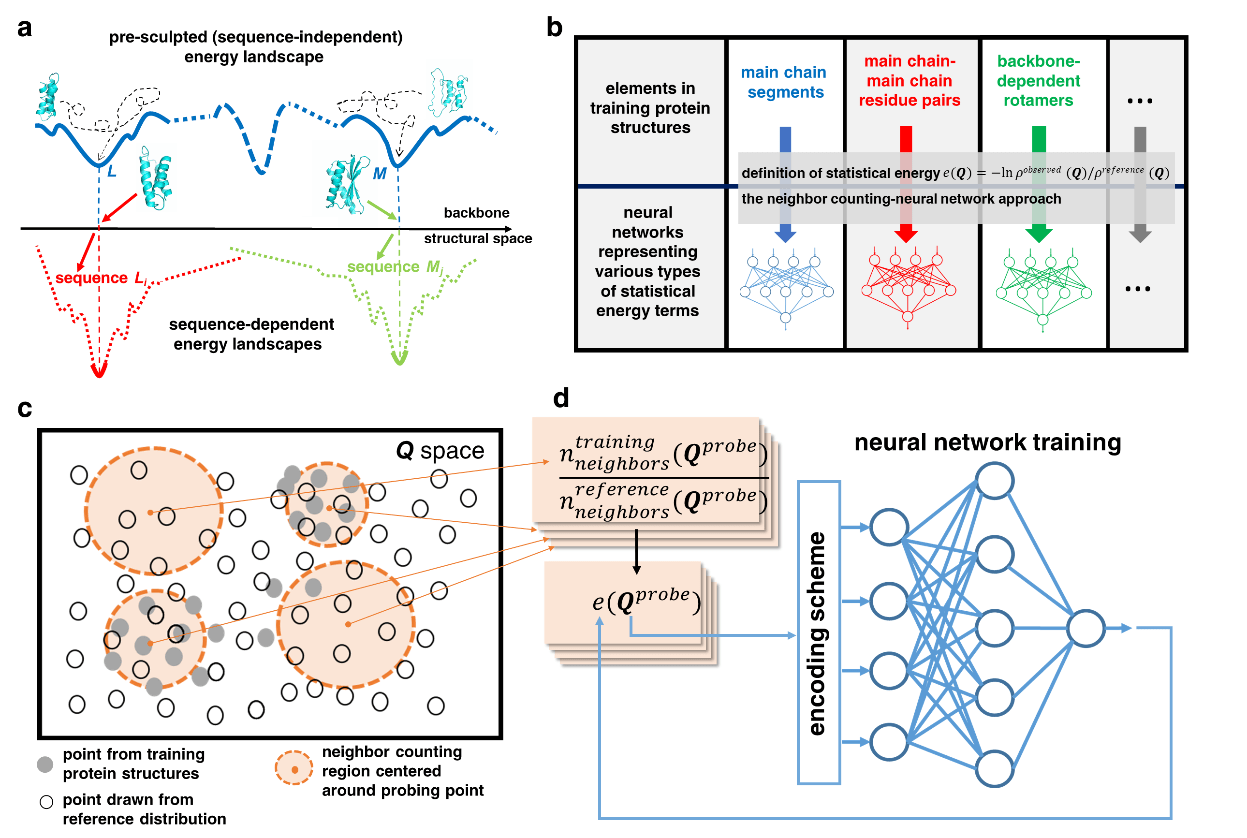
▲ 图 1.用 SCUBA 模型进行蛋白质设计的原理 。 (a) SCUBA 主链能量面上的极小对应了蛋白质的可设计主链结构 , 即特定氨基酸序列下的最低自由能结构;(b) SCUBA 中用神经网络表示的统计能量项;(c) 和(d) 用近邻计数(NC)-神经网络(NN)方法从蛋白质结构原始数据中学习解析能量函数的方法框架 | 图源:中国科大官网
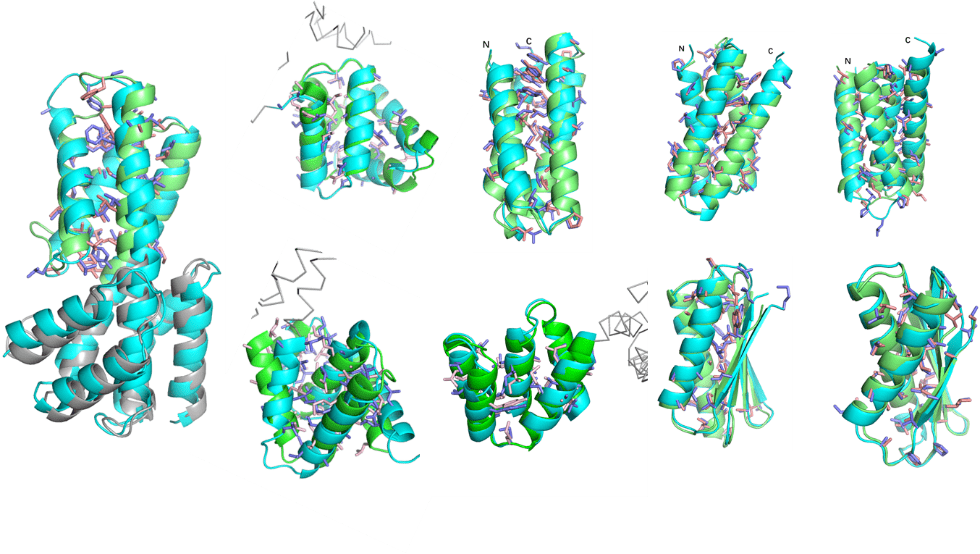
理论计算和实验证明 , 用 SCUBA 设计主链结构 , 能够突破只能用天然片段来拼接产生新主链结构的限制 , 显著扩展从头设计蛋白的结构多样性 , 进而设计出不同于已知天然蛋白的新颖结构 。 “SCUBA 模型 + ABACUS 模型”构成了能够从头设计具有全新结构和序列的人工蛋白完整工具链 , 是 RosettaDesign 之外目前唯一经充分实验验证的蛋白质从头设计方法 , 并与之互为补充 。 在论文中 , 团队报道了 9 种从头设计的蛋白质分子的高分辨晶体结构 (图 2) , 它们的实际结构与设计模型一致 , 其中 5 种蛋白质具有天然蛋白质中尚未观察到的新型拓扑结构 。
文章图片
▲ 图 2.从头设计蛋白的高分辨晶体结构(天蓝色)与设计模型(绿色)比较 | 图源:中国科大官网
https://www.nature.com/articles / s41586-021-04383-5
推荐阅读
- 序列|我国科学家建立蛋白质设计新方法
- 全套|佳明本能Instinct 2系列来袭!个性化设计、超大电力续航...还有全套智能监测
- Apple|苹果推出无面盖的C222x磁吸无线充电模组 解锁个性化设计
- 设计|推进商业数字化转型 促进消费提质扩容(消费万花筒)
- 设计|徕卡发布两款腕表产品 售价99800元
- 硬件|Razor与Jeep合作推出RX200滑板车 融入Wrangler的粗犷细节设计灵感
- Xiaomi|南芯半导体声明:小米澎湃P1为小米自研设计
- 强劲|诺基亚N8复刻版曝光,骁龙8 gen 1+强劲四摄,经典设计重现
- 设计|有品众筹上线Lydsto自动集尘手持无线吸尘器,众筹价1299元
- 设计|新年开工新装备,这六款“香到爆”华为笔记本,助你成为职场达人